邬建敏课题组在组织分子质谱成像领域取得进展
背景介绍
分子病理作为跨界融合的技术逐渐成为精准医疗的支柱,其在疾病的鉴别诊断、伴随诊断、分子分型、预后判断等方面起到极其重要的作用。目前,分子病理学中常用的技术包括高通量基因测序(NGS),荧光原位杂交(FISH),免疫组化(IHC)、基因芯片及PCR技术等。随着精准医学的发展,对复杂疾病尤其是恶性肿瘤等疾病的个性化精准治疗需要从更多维度的分子角度对病理组织样本进行分型和判别。由于质谱技术的高通量,免标记特点,质谱成像、质谱组织活检、流式细胞质谱等技术将成为分子病理领域的重要方向。
脂质与细胞增殖、细胞物质交换与能量交换、细胞信号传导等重要生理生化过程密切相关。基于空间脂质组学的分子病理研究对于推动精准医学的发展具有重要意义。质谱成像是空间脂质组学的主要解决方案。其中,基质辅助激光解吸离子化质谱成像( MALDI-MSI)是应用最为广泛的质谱成像技术,但由于有机小分子基质的背景干扰、结晶不均匀性及前处理时间长等缺点使得常规MALDI-MSI在临床应用中受到阻碍。化学系邬建敏课题组此前在免基质脂质分析领域开展了一系列的研究。该课题组利用垂直硅纳米线(SiNW)顶端的电磁增强效应,提出了顶端接触取样原位离子化质谱技术(TCSI-MS),实现了组织样本脂质分子的快速获取(ACS Appl. Mater. Interfaces,2018,10, 14389).与附属邵逸夫医院合作成功地将TCSI-MS技术应用于肝癌组织样本的良恶性判断和切缘判别 (Anal. Chem. 2019,91, 10376)。TCSI-MS技术通过组织与纳米线顶端的接触,将组织脂质分子转印到纳米线,在后续的质谱分析中,激光轰击纳米线顶端进而实现组织样本分子的无损分析。该方法支持同一个组织样本的跨平台分析,便于获取多组学分子信号。然而,因硅纳米线阵列表面的空间不均匀性,TCSI-MS在组织成像、空间代谢组或脂质组学的应用仍然面临挑战。发展一种能高保真呈现脂质分子在组织表面空间分布的免标记成像基板是解决TCSI-MS在分子病理领域应用的关键。
近日,化学系邬建敏课题组在ACS Nano上发表了基于组织转印技术的质谱成像新方法,实现了TCSI-MS在组织分子成像领域的应用。
工作亮点
研究人员首先使用自下而上生长的两亲性氮掺杂石墨烯量子点(NGQD)作为插层剂和分散剂来辅助MoS2的液相剥离,以获得薄而稳定的NGQD@MoS2二维纳米片。然后通过自组装过程将它们负载到硅纳米线(SiNWs)的顶端,获得一种新型的NGQD@MoS2/SiNWs质谱成像基板(图1)。
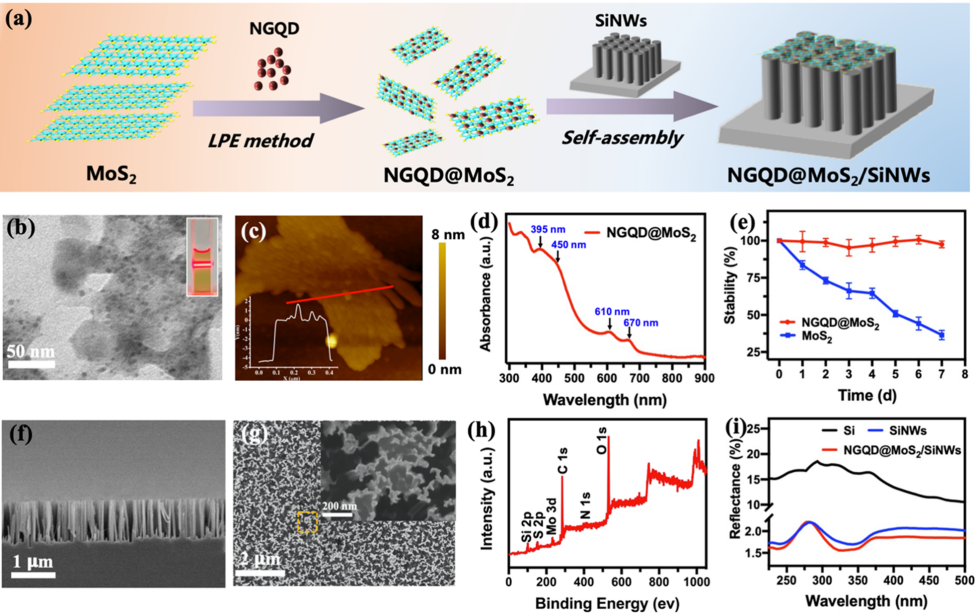
图 1. (a)NGQD@MoS2/SiNW基板合成流程示意图;(b)NGQD@MoS2纳米片TEM图像;(c)NGQD@MoS2纳米片AFM图像;(d)NGQD@MoS2纳米片紫外吸收曲线;(e)NGQD@MoS2与MoS2纳米片稳定性比较;(f)NGQD@MoS2/SiNW截面SEM图像;(g)NGQD@MoS2/SiNWs表面 SEM图像;(h)NGQD@MoS2/SiNW XPS谱图;(i)Si,SiNWs, NGQD@MoS2/SiNWs紫外漫反射曲线。
在脂质标准品测试中,通过与MoS2/SiNWs、NGQD/SiNWs、SiNWs基板对比,NGQD@MoS2/SiNWs基板显示出优异的LDI性能(图2 a-f)。同时为了验证基板表面是否能得到均一的质谱信号(或“sweet-spots"效应的消除),研究人员扫描了整个脂质标品液滴干燥沉积后的区域进行质谱成像,发现NGQD@MoS2/SiNWs基板的信号更强且更为均匀(图2g)。
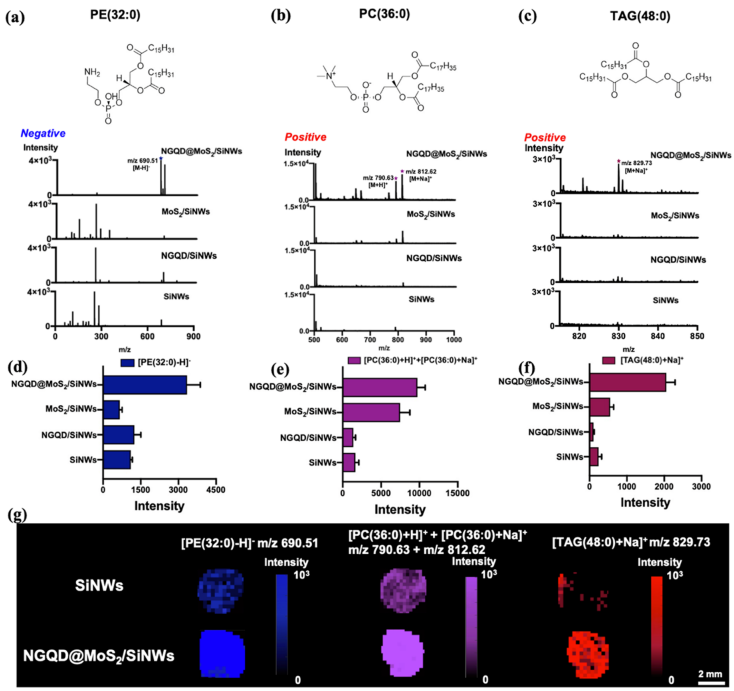
图 2 (a)-(c)脂质分子结构式及代表性谱图;(d)-(f)脂质分子在不同基板上的出峰强度对比;(g)脂质分子在不同基板上沉积区域的成像扫描图。
为研究复合材料的协同作用机制,研究人员用靛蓝和苄基吡啶盐作为模型分子探究了不同基板的表面光致电荷转移能力、热解吸及内能转换效率。从靛蓝分子负离子模式下的总离子强度(图3b)可以看出,NGQD@MoS2/SiNWs基板比SiNWs基板高出40倍,也远远高于MoS2/SiNWs及NGQD/SiNWs基板。这可能是由于NGQD@MoS2具有超高的载流子寿命,而NGQD有益于促进电荷转移并有效抑制光致电子-空穴对的复合。载流子寿命的明显提升及SiNW-MoS2间P-N异质结的共同作用,使得光致电荷转移几率得到极大的提升(图3d)。通过对苄基吡啶盐的检测,发现NGQD@MoS2与SiNWs复合后可将苄基吡啶盐母离子存活率从54%提升到71%(图3g),这是由于NGQD@MoS2纳米片可以具有sp2结构,与SiNWs复合后,能量可以分散到sp2结构中,从而降低内部能量转移,减少分子碎片,有助于LDI能力的提升。
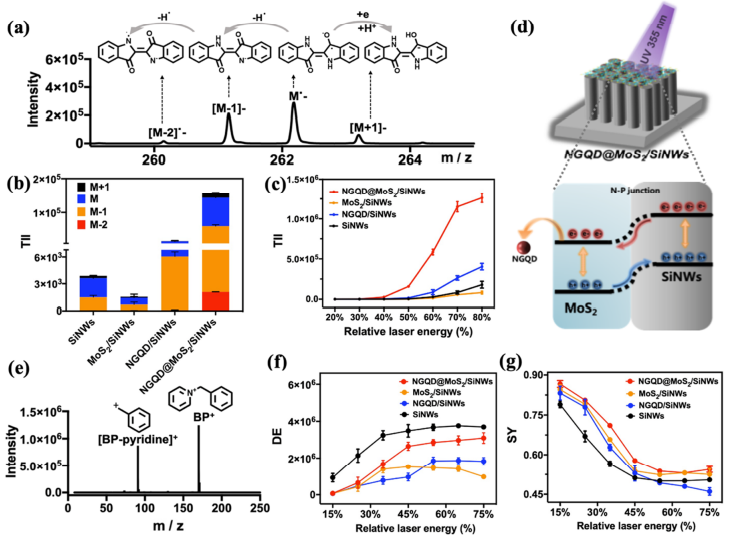
图 3.(a)靛蓝分子在负离子模式下的谱图;(b)-(c)靛蓝分子总离子强度对比图;(d)协同机理示意图;(e)苄基吡啶盐正离子模式谱图;(f)苄基吡啶盐总离子强度对比图;(g)存活率曲线。
得益于NGQD@MoS2/SiNWs基板的高LDI效率和优异的分子成像能力,研究人员成功地对小鼠肺组织进行了印迹质谱成像。在NGQD@MoS2/SiNWs上获得的脂质数量和总离子强度比在 SiNWs基板上高 4-5倍。成像结果可清晰显示小鼠肺组织的脂质分子空间分布和空泡孔洞结构(图 4)。此基板可兼容印迹采样和成像的功能,具有抗干扰能力强、灵敏度高等优点。该方法将质谱成像前处理流程缩短至10分钟以内,更有利于与临床医学研究相结合。
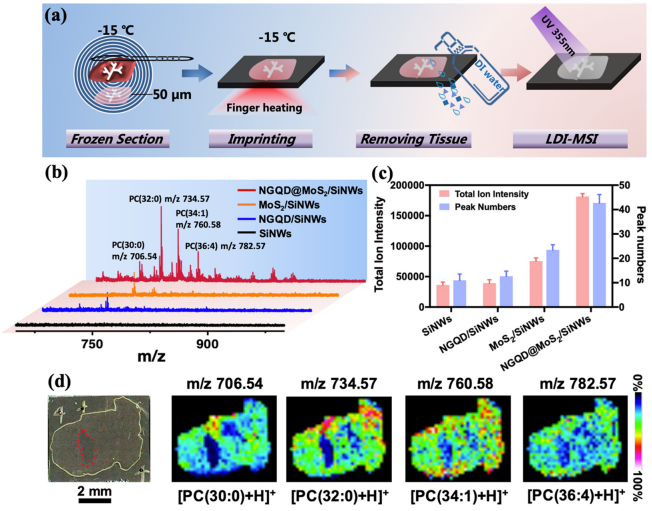
图4. (a)印迹质谱成像流程;(b-c)不同基板获得的成像效果对比;(c)小鼠肺组织质谱成像图。
基于此项技术,研究人员对非小细胞肺癌(NSCLC)脂质代谢生物标志物进行了挖掘和可视化研究分析。通过对30例肺癌组织及相邻正常组织进行脂质组学分析,发现了7个脂质分子可作为NSCLC的生物标志物。为了验证这些生物标志物的可靠性,研究人员进行了受试者特性曲线(ROC)分析。结果显示,单一的脂质分子作为标志物时的曲线下面积 (AUC)值范围为 0.73-0.95(图 5c)。通过7个脂质特征分子的标志物组组合所得AUC值 为 0.982(图 5d),表明上述脂质分子构成的诊断Panel对NSCLC组织和相邻组织具有极高的判别准确率。
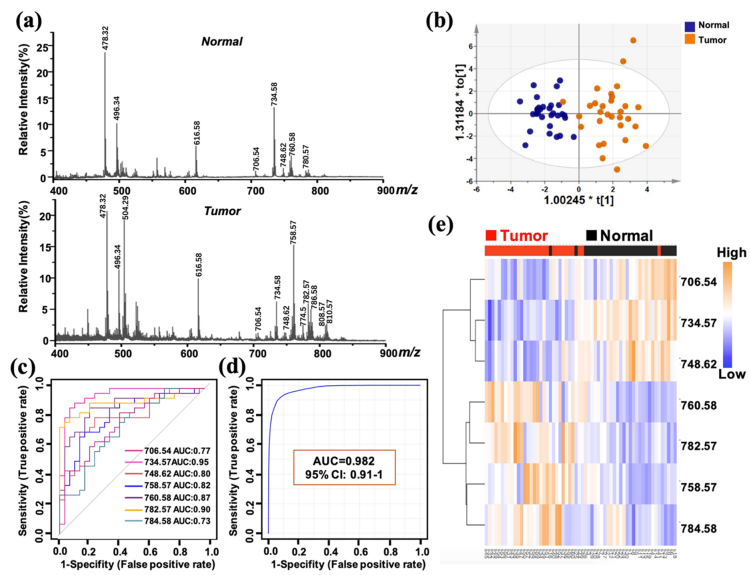
图 5. (a)肺癌癌症组织(Tumor)及其相邻正常组织(Normal)成像后得到的平均谱图;(b)OPLS-DA分数图;(c)单一生物标志物ROC曲线及AUC值;(d)生物标志物组ROC曲线及AUC值;(e)层状聚类图。
质谱成像图像在可清晰呈现上述7个特征脂质分子在肿瘤组织和相邻正常组织的空间分布差异。这一结果显示了TCSI-MSI在基于脂质组学的肿瘤组织诊断方面的独特的优势,有望推动脂质组学在精准医学中的发展。
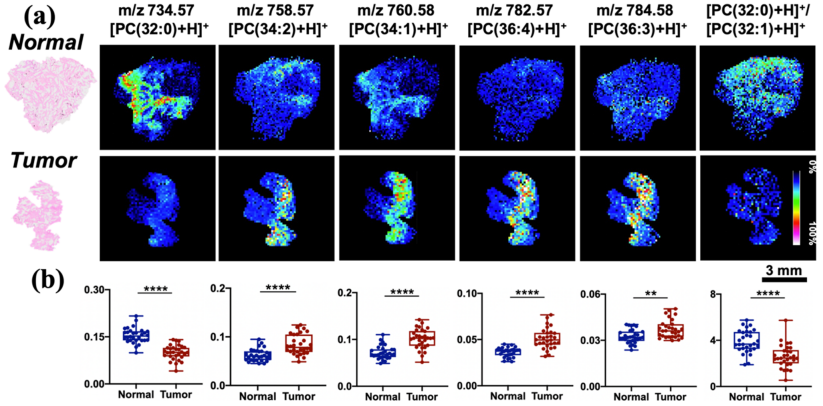
图6. 肺癌癌症组织(Tumor)及其相邻正常组织(Normal)HE染色图像及不同脂质分子的质谱成像图。
论文的第一作者是化学系19级博士生刘星月。本论文采用的NGQD辅助液相剥离MoS2技术与浙江理工大学奚凤娜(共同通讯作者)课题组合作;论文的临床研究与附属邵逸夫医院何正富主任(共同通讯作者)和陈钊医生(共同一作)合作。本课题的研究工作受国家自然科学基金(21874118)和杭州汇健科技有限公司的资助。
原文链接:https://pubs.acs.org/doi/full/10.1021/acsnano.2c02616

